温馨提示:请使用火狐或者Chrome的网页浏览器来查看报告
微科盟转录组和代谢组关联分析云流程结题报告
一 概述
转录组、代谢组等单个组学分析技术都通过单变量统计方法(包括方差分析、线性模型或 t 检验)进行独立分析。 然而,不同组学协同作用以调节和影响生物系统和信号通路,所以单一组学分析忽略了不同组学之间的关系,可能会遗漏关键的生物学信息[1]。 为了更系统和全面地探索和理解这些复杂的机制,需要使用多组学关联分析的方法整合多组学数据,在转录组和代谢组两个水平共同研究生物学问题,筛选关键基因/代谢物,深入解释宏观的生物学现象,从而在两种系统水平全面地理解复杂的生物学[2]。
二 项目流程
转录组和代谢组关联分析云流程包含了以下数据分析方法。
结合近期相关的研究,在本云流程中从以下三个方面实现了转录组和代谢组的关联分析。
- 1,基于相关性的关联分析。在该部分实现了转录组和代谢组的相关性热图,九象限图和相关性网络图。
- 2,基于KEGG(Kyoto Encyclopedia of Genes and Genomes)通路和KEGG注释的富集分析[3]。在该部分实现了基于KEGG
ORA和KEGG GSEA的富集分析,基于组合概率检验(p Value Combination Methods)的共富集分析和KEGG上色通路图。
- 3,基于多元统计学和统计模型的关联分析。在该部分实现了典型相关分析(Canonical Correlation Analysis,CCA), 偏最小二乘分析(Partial Least Squares
Analysis, PLS),PLS-DA分析(Partial Least Square-Discriminant Analysis)和双向偏最小二乘分析(Two-way Orthogonal Partial
Least Squares Analysis,O2PLS)。
三 数据分析结果
在该部分为您简述云流程涉及到的方法及分析结果。按照统计学原理将所有分析分为三类:1,基于相关性的关联分析;2,基于KEGG数据库的共富集分析和通路图;3,基于多元统计学和统计模型的关联分析。
3.1 基于相关性的分析
3.1.1 相关性热图
热图可以聚合大量的数据,并很直观地展示基因和代谢物的相关性。本项目结果图见结果文件
01_相关性热图/本部分包含以下结果:
├── 01_相关性热图
├── cor_p.csv [p Value值,您可以将其导入Excel查看并编辑]
├── cor_r.csv [该表为Spearman相关分析的]相关系数值,您可以将其导入Excel查看并编辑]
└── result.svg [相关性热图]
图3.1 转录组和代谢组的相关性热图
注:在该图中,行代表代谢物而列代表基因。默认使用Spearman法计算每个基因和代谢物的相关性,默认筛选30个基因和代谢物并用行表示基因用列表示代谢物。在热图中以不同颜色代表相关系数值R,红色代表基因表达量和代谢物丰度具有正相关关系,蓝色代表负相;饱和度代表相关关系的强弱,颜色越饱和代表相关关系越强;对于具有统计学上的显著差异的相关关系,用星号进行标注。若p Value<=0.05,则表示为*,若p Value <=0.01,则表示为**。
3.1.2 九象限图
转录组和代谢组的九象限图能够反应差异基因与差异代谢物之间的关系。可在分步骤流程中对图形参数进行更进一步的设置。本项目结果图见结果文件
02_九象限图/本部分包含以下结果:
├── 02_九象限图
├── outputData.tsv [基因与代谢物相关性数据]
└── result.html [动态网络图]
图3.2 转录组和代谢组的九象限图
注:九象限图可以表示差异表达基因和差异代谢物的关系。x轴表示两个处理间基因的变化倍数(经过log2变换),y轴表示两个处理间代谢物的变化倍数(经过log2变换)。黑色虚线代表不同的阈值并将图划分为九个象限,按照从左到右从上向下分别编号为象限1-9;每个点表示一个基因和一个代谢物的相关关系。使用鼠标悬停在点上可以显示点所对应的基因和代谢物各自的ID,log2Fold
Change,及其相关系数,p Value和所在象限数。
九象限图可从两方面进行解析。1,按照两个处理基因和代谢物差异是否显著,第5象限的点所对应的基因表达量和代谢物丰都差异均不显著;第2,8象限的点代表两个处理的基因表达量差异不显著而代谢物的积累情况变化显著;第4,8象限的点代表代谢物丰度差异不显著而基因的表达情况变化显著;1,3,7,9象限代表代谢物积累和基因表达情况均变化显著。2,按照转录组和代谢组表达量变化的高低,1,2,4象限代谢物的变化倍数高于基因的;3,7象限基因和代谢物具有相同的变化模式;6,8,9象限基因的变化倍数高于代谢物。
3.1.3 相关性网络图
对两个具有网状相关关系的组学数据集进行建模的一个简单的方式是使用相关性网络图(Relevance Networks)[4]。在相关性热图中,节点代表基因或代谢物,用不同形状表示;而边代表基因和代谢物之间的联系。 由于绘制同种组学(基因-基因之间或代谢物-代谢物之间)相关性网络图的工具众多,在本流程中只关注基因与代谢物之间的相关性,即只描述基因与代谢物之间的相关性。
相关性网络图具有以下优势:
- 1,可以同时表示正相关和负相关关系;
- 2,能够同时反应不同通路之间基因与代谢物的关系[5];
- 3,相关性网络图还能够直观的显示具有最高连接度的基因(Hub Genes)或代谢物,这类基因或代谢物通常具有相对重要的生物功能。您可以结合相关生物学研究进行进一步分析。
本项目结果图见结果文件03_相关性网络图/
本部分包含以下结果:├── 03_相关性网络图
└── result.html
图3.3 相关性网络图
注:使用交互式图展示基因和代谢物的相关性网络图。在相关性网络图中,分别用圆形节点和方形节点代表基因和代谢物。用边表示对于符合筛选条件(默认为相关系数绝对值大于0.8,p Value小于0.05)的基因与代谢物的关系;并在图中展示关键基因和代谢物(默认为前10%)的ID。使用鼠标悬停在节点上可查看基因或代谢物信息(节点ID,类型,连接度),悬停在边上可以查看相关性信息(该相关关系对印的基因、代谢物及相关系数)。 可在分步骤流程中对图形参数进行更进一步的设置及导出静态图片。
3.2 基于KEGG的分析
与基于单个基因/分子的方法相比,通路分析更灵敏、更一致、信息更丰富[6]。KEGG(Kyoto Encyclopedia of Genes and Genomes) 数据库是一个整合了基因组信息与高等级功能信息的数据库[3],是基因和基因组的百科全书,其被广泛运用于各种组学的研究中。本部分分析能够将您的组学数据与KEGG数据库结合,结果有助于更加深刻的揭示不同组学水平的变化及其生物学意义。
KEGG数据库包含15个依据前人发表的文献手动整理的部分[7]。如表3.1所示:
| 分类 | 数据库名称 | 内容 | KEGG标识符 |
|---|---|---|---|
| 系统信息 | KEGG PATHWAY | 代谢通路图 | Map |
| 系统信息 | KEGG BRITE | BRITE 层次结构和表 | br/ko |
| 系统信息 | KEGG MODULE | 模块 | M |
| 基因组信息 | KEGG ORTHOLOGY(KO) | 直系同源基因 | K |
| 基因组信息 | KEGG GEONME | 物种基因组(包括一部分病毒基因组) | org code/T number |
| 基因组信息 | KEGG GENES | KEGG生物、病毒和附录类别的基因目录 | org:gene |
| 基因组信息 | KEGG SSDB | 条目之间的序列相似性 | |
| 化合物信息 | KEGG COMPOUND | 代谢物或其他小分子 | C number |
| 化合物信息 | KEGG GLYCAN | 多糖 | G number |
| 化合物信息 | KEGG REACTION | 生化反应 | R number |
| 化合物信息 | KEGG RCLASS | 反应类 | RC number |
| 化合物信息 | KEGG ENZYME | 酶的命名 | EC number |
| 健康信息 | KEGG DISEASE | 疾病 | H number |
| 健康信息 | KEGG DRUG | 药物 | D number |
| 健康信息 | KEGG DGROUP | 药物分类 | DG number |
| 健康信息 | KEGG ENVIRON | 天然药物和健康相关物质 | E number |
表3.1 KEGG的不同数据库类型
(资料来源:Kanehisa M., Furumichi M., et al. KEGG: New Perspectives on Genomes, Pathways, Diseases and Drugs. Nucleic Acids Res. 45, D353–D361 (2017).)
这里重点介绍与转录组和代谢组关联分析相关的以下三个数据库:KEGG ORTHOLOGY,KEGG COMPOUND和KEGG PATHWAY。
- 1, 直系同源(Orthology)是指来源于被比较物种的最后一个共同祖先(last common ancestor, LCA)中同一个祖先基因的基因,随着物种形成事件(Speciation
Events),祖先基因被不同的物种分别继承。由于具有同源性,直系同源基因在功能上往往是一致的,但并不绝对[8]。KEGG
ORTHOLOGY数据库(KO)包含具有直系同源关系的基因(或编码蛋白质)信息及其所对应的功能。KO数据库中,直系同源基因以K开头。
- 2, KEGG
COMPOUND数据库主要存储着小分子、生物聚合物和其他与生物系统相关的化学物质(相对分子质量通常在1000以下)。在该数据库中的代谢物以C开头,例如C00031为葡萄糖。
- 3, KEGG
PATHWAY是KEGG数据库中最主要的部分,它储存着手动绘制的参考通路图。这些参考通路图由不同的物种特异性通路图依据基因间的直通同源关系计算生成,其中矩形代表直系同源基因而圆形代表代谢物。
每个通路的总图为map开头+五位数字编号(例如Map00020为TCA循环的总图),而物种代码+五位数字编号组成物种特异的通路图(例如hsa00020为人类的TCA循环通路。其中人类特有的直系同源基因用绿色表示)。
需要注意以下两组特殊的通路:Map01100(全局通路图)和Map01200(总览图),这两个通路分别代表代谢物的总体特征和主要特征,故不包含直系同源基因(矩形)。
3.2.1 共富集分析
高通量测序技术的发展使得转录组、代谢组及其他各种组学均能产生大量数据,然而仅依靠不同样本间的表达量信息难以挖掘出潜在的生物学意义。其中一种解决方法是将大量差异基因或蛋白质按照功能进行分组,对不同的分组进行分析,从而降低分析的复杂性[9]。在分组时可以使用已知的信息,例如GO,KEGG等数据库。这种类分析方法统称为富集分析。多种方法可以实现富集分析。这里为您介绍最常见的两种实现富集分析的方法:
过代表分析(Over-Representation Analysis, ORA)使用四格表法[10]并大致遵循以下策略:1,需要用特定阈值或者条件筛选出差异表达基因(或代谢物);2,统计每个通路中差异表达基因的数目;3,依次检测每个通路中差异基因占全部基因的比例是否显著高于全部差异基因占背景基因总数的比例,如果某条通路差异基因的比例显著高于背景基因集,则推断该通路整体存在差异表达。比较两者比例是否有差异可使用超几何分布检验、卡方检验或二项分布检验等统计方法。
然而,该方法至少存在以下局限:1,该方法只能平等的对待每个发生差异表达的基因(或代谢物),而无法考虑表达量变化情况。2,该方法无法考虑没有被筛选出发生差异表达的基因(或代谢物),使用这种方法,会遗漏相对不重要的基因/代谢物(例如,Fold Change = 1.999 或 p Value = 0.051的基因/代谢物),从而导致信息丢失。为了克服这些不足,功能类评分(Functional Class Scoring,FCS)方法被用于富集分析。该方法统计每个基因的表达量变化情况,然后将每个通路上的所有基因表达量变化情况整合成一个统计量并对这个统计量进行假设检验。基因集富集分析(Gene Set Enrichment Analysis,GSEA)是一个被广泛使用的FCS富集方法[11]。
R语言中的clusterProfiler包提供了丰富的富集分析功能[12],本云流程使用clusterProfiler进行KEGG ORA和KEGG GSEA富集分析。
转录组、代谢组等单一的组学富集分析只会从特定水平解释生物信息,共富集分析能够整合转录组和代谢组富集分析,从而能更全面、更系统地揭示生物过程的重要部分。本云流程中整个转录组和代谢组富集分析结果参照了multiGSEA软件包[13]中整合富集分析结果的统计方法,包括Fisher组合概率检验(Fisher's Combined Probability Test),Stouffer's Method和Edgington's Method等并使用poolR[14]包重写了相关代码,使其可以避免multiGSEA软件包的物种限制,同时使整合方法可以运用于KEGG ORA富集分析。这里选择最常见的Fisher组合概率检验进行简要描述。
对于每一个通路转录组和代谢组的富集结果p Value,构建如下统计量: $$\chi_F^2 = -2 \sum_{i=n}^k \ln(p_i)$$ 其中,k为独立检验的个数,在本云流程为2(转录组和代谢组);p分别代表该通路每个组学的p Value。该统计量服从自由度为\(2^k\)的\(\chi^2\)分布。因此使用卡方检验便可以计算整合p Value。
共富集分析显著的KEGG通路会以表格(表3.2)的形式进行展示。本项目结果图见结果文件 04_共富集分析/
本部分包含以下结果:
├── 04_共富集分析
├── combinationTest.tsv [共富集分析结果]
├── enrich_ora_cpd.tsv [代谢组富集结果]
├── enrich_ora_gene.tsv [转录组富集结果]
├── metabenrich_analysis_ora.svg [代谢组富集条形图]
└── transenrich_analysis_ora.svg [转录组富集条形图]
| mapID | Description | transcriptomePvalue | metabolomePvalue | combinationPvalue |
|---|---|---|---|---|
| 02010 | ABC transporters | 0.000830 | 1.000000 | 0.006719 |
| 04210 | Apoptosis | 0.002321 | 1.000000 | 0.016397 |
| 04514 | Cell adhesion molecules | 0.003897 | 1.000000 | 0.025518 |
| 00591 | Linoleic acid metabolism | 1.000000 | 0.000142 | 0.001404 |
| 00590 | Arachidonic acid metabolism | 1.000000 | 0.000183 | 0.001755 |
表3.2 共富集分析结果
注:mapID:富集到的通路ID;Description:富集到通路的简短描述;transcriptomePvalue:转录组数据富集到该通路的p Value;metabolomePvalue:代谢组数据富集到该通路的p Value;combinationPvalue:整合p Value。
3.2.2 联合通路图
基于KEGG的联合通路图可以反映同一通路内基因和代谢物变化情况。本项目结果图见结果文件 05_联合通路图/
本部分包含以下结果:├── 05_联合通路图
├── *.png [图片版通路图]
├── *_result.html [网页版通路图]
图3.4 转录组和代谢组的联合通路图
注:在联合通路图中,不同类型的节点代表不同对象:矩形代表基因的表达产物(通常为蛋白质但少量为RNA);圆形代表代谢物,DNA或其他分子;圆角矩形代表通路。对象之间的关系用不同类型的箭头标注:黑色实线箭头代表分子间反应或关系;白色实线箭头代表联系到其他通路;黑色虚线箭头代表间接或未知反应,等等。更进一步的说明请查阅Regular Map Notation。通路中差异基因或代谢物会按照分组以热图形式展示表达量或丰度并在图片右上角标注图例。您可以与通路图交互,使用鼠标悬停在节点上可以显示对象ID,点击对象可以连接到KEGG网站详情页。
3.3 多元统计模型分析
基于多元统计(降维、潜变量)的方法提供了更好的对转录组和代谢组数据进行探索性分析,能够突出显示两组学之间的全局相关性,以及找出单个组学数据中的异常值或批次效应,还有助于处理和基因/代谢物的下游分析[15]。这里对本流程中的涉及的多元统计分析方法(包括CCA, PLS, PLS-DA, O2PLS)进行简要描述。
为了使表达一致,首先对不同文献中的公式符号记法进行统一。在本节出现的公式中进行如下约定:大写加粗的字母(例如\(\boldsymbol{X}\))表示矩阵,小写加粗的字母(例如\(\boldsymbol{f}\))表示向量,而小写不加粗的字母(例如\(n,p\))表示标量。
许多数学对象可以通过将它们分解成多个组成部分或者找到它们的一些通用的属性来更好的理解。给定一个组学数据集 \(\boldsymbol{X}\),它是一个\(n \times p\)矩阵,由\(n\)个观测值(也就样本数)和\(p\)个变量(也就是基因或代谢物的数量)组成,它可以用以下形式表示: $$\boldsymbol{X} = (\boldsymbol{x}_1,\boldsymbol{x}_2,\cdots,\boldsymbol{x}_p)$$ 注意,该式中每个\(\boldsymbol{x}\)都是一个\(n\)维向量,包含了\(n\)个样本的测量值(基因表达量或代谢物丰度)。大多数情况下,组学研究的变量数远大于样本数量\(p>>n\),也就是基因/代谢物的数量远大于样本数。我们需要找到一组新的变量,这些新的变量由原始数据的线性组合组成并且能够捕获原始数据中的绝大部分方差。通常,这些变量被称为隐变量(Latent Variable)或被称成分(Component)。其中一个隐变量\(\boldsymbol{f}\)可以由以下公式表示: $$\boldsymbol{f}=q_1 \boldsymbol{x}_1+q_2 \boldsymbol{x}_2+\cdots+q_p \boldsymbol{x}_p$$ 此处,\(\boldsymbol{f}\)是\(n\)维向量。该式也可以转化为矩阵向量乘法的形式: $$\boldsymbol{f} = \boldsymbol{X}\boldsymbol{q}$$ 该式中系数向量\(\boldsymbol{q} = (q_1,q_2,\cdots,q_p)^T\)控制原始组学数据集的每个变量的缩放,被称作载荷(loading)。隐变量的数量远远小于\(p\)。通过将原始组学数据矩阵分解的形式,少量的隐变量即能够描述整个组学数据。
3.3.1 CCA分析
对一组数据降维的方法可以拓展到多组数据。包括协惯量分析(Co-Inertia Analysis, CIA),偏最小二乘分析(Partial Least Squares, PLS),典型对应分析(Canonical Correspondence analysis, CCA)和典型相关分析(Canonical Correlation Analysis , CCA)等方法。需要注意的是典型对应分析和典型相关分析的英文缩写均为CCA。典型对应分析是对应分析(Correspondence analysis, CA)的一种约束形式,它广泛应用于生态学方向的研究却并未广泛运用于多组学关联分析[15]。本流程用CCA表示典型相关分析。
在CCA分析中,两个组学数据集 \(\boldsymbol{X}\)(维度为 \(n \times p_1\)) 和\(\boldsymbol{Y}\)(维度为 \(n \times p_2\))可以用以下隐变量分解的形式表示: $$\boldsymbol{X}=\boldsymbol{F}_x \boldsymbol{Q}_x^T+\boldsymbol{E}_x\\\boldsymbol{Y}=\boldsymbol{F}_y \boldsymbol{Q}_y^T+\boldsymbol{E}_y $$ 式中\(\boldsymbol{F}_x\)和\(\boldsymbol{F}_y\)为维度为\(n \times r\)维矩阵,该矩阵包含r个的隐变量(成分),每个隐变量是n维向量。隐变量矩阵\(\boldsymbol{F}_x\)和 \(\boldsymbol{F}_y\)解释了两个组学数据的共同结构(co-structure)。\(\boldsymbol{Q}_x\) 和\(\boldsymbol{Q}_y\)是\(\boldsymbol{F}_x\) 和 \(\boldsymbol{F}_y\)的载荷,而\(\boldsymbol{E}_x\)和\(\boldsymbol{E}_y\)代表误差项。CCA分析通过最大化\(\boldsymbol{Xq}_x^i\)和\(\boldsymbol{Yq}_y^i\)的相关性来获得组学数据集 \(\boldsymbol{X}\)和\(\boldsymbol{Y}\)的相关性。
然而在进行CCA分析时,由于需要计算隐变量相关性矩阵的逆矩阵,因此无法直接在变量数远大于样本数量的组学数据集的分析中使用[16]。一些CCA分析的变体可以解决上述问题从而使其可以使用在两个组学的关联分析中。例如Sparse CCA(sCCA)[1],[17]来进行多组学关联分析。 本项目结果图见结果文件 06_CCA分析/
本部分包含以下内容:├── 06_CCA分析
├── arrow.svg [转录组和代谢组隐变量关联散点图]
├── biplot.svg [双序图]
├── plotVar.svg [相关性圈图]
└── scatter.svg [载荷图]
(a)
(b)
(c)
(d)
图3.5 CCA分析
注:(a)CCA分析双序图,该图具有两套原点和单位向量方向相同的坐标系。图中散点代表隐变量而箭头代表载荷。散点表示样本在PC1和PC2轴上的隐变量的贡献度,对应Component坐标系(左下坐标轴);从原点出发的箭头的端点表示载荷值,对应Eigenvector of Component坐标系(右上)。其生物学意义的解析见结题报告3.3.5 重要的图的解读;(b)隐变量散点图,其中Block1和Block2分别代表转录组和代谢组,散点表示样本在PC1和PC2轴上隐变量的贡献度;(c)同一隐变量对转录组和代谢组分别的贡献;(d)相关性圈图,代表载荷的分布情况。3.3.2 PLS分析
PLS是高维组学数据分析中的另外一种高效降维方法。同CCA类似,PLS也使用隐变量分解的方式对两组学数据集降维并寻找其共同结构,但PLS最大化协方差矩阵而不是隐变量间的相关性[18]。这种方式使得PLA的结果更加稳健并能够更好的处理极端值的影响,同时,PLS法不会像CCA一样遭遇\(p≫n\)时无法求解的问题。本项目结果图见结果文件 07_PLS分析/
本部分包含以下内容:├── 07_PLS分析
├── arrow.svg [转录组和代谢组隐变量关联散点图]
├── biplot.svg [双序图]
├── plotVal.svg [相关性圈图]
└── scatter.svg [载荷图]
(a)
(b)
(c)
(d)
图3.6 PLS分析
注:(a)PLS分析双序图,该图具有两套原点和单位向量方向相同的坐标系。图中散点代表隐变量而箭头代表载荷。散点表示样本在PC1和PC2轴上的隐变量的贡献度,对应Component坐标系(左下坐标轴);从原点出发的箭头的端点表示载荷值,对应Eigenvector of Component坐标系(右上)。其生物学意义的解析见结题报告3.3.5 重要的图的解读;(b)隐变量散点图,其中Block1和Block2分别代表转录组和代谢组,散点表示样本在PC1和PC2轴上隐变量的贡献度;(c)同一隐变量对转录组和代谢组分别的贡献;(d)相关性圈图,代表载荷的分布情况。3.3.3 O2PLS分析
不同类型的组学研究在规模,分布形状,实验误差等方面存在差异,而上文中提到的CCA和PLS并为考虑不同组学特有的变异。在构建模型时将不同组学特有的变异纳入模型,可能会使得模型的性能更好[19]。O2PLS算法正是基于这种思想被提出。
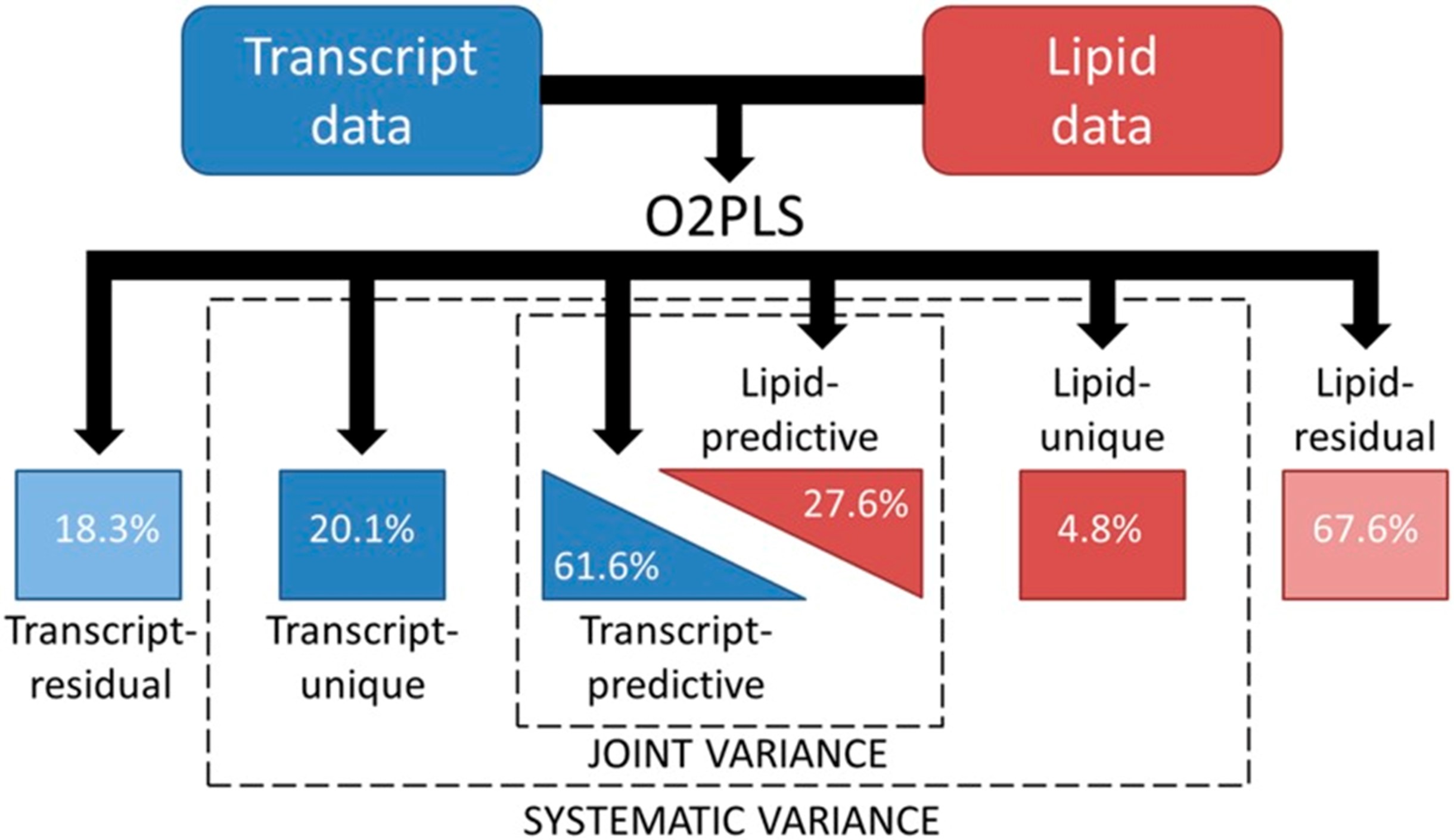
图3.7 O2PLS算法的示意图
(资料来源:Szymanski J., Brotman Y., et al. Linking Gene Expression and Membrane Lipid Composition of Arabidopsis. Plant Cell 26, 915–928 (2014).)
在O2PLS模型中,两个组学数据集\(\boldsymbol{X}\)(维度为 \(n \times p_1\)) 和\(\boldsymbol{Y}\)(维度为 \(n \times p_2\))可以用以下隐变量分解的形式表示: $$\boldsymbol{X}=\boldsymbol{TW}^T+\boldsymbol{T}_{Y⊥}\boldsymbol{p}_{Y⊥}^T+\boldsymbol{E}_x\\\boldsymbol{Y}=\boldsymbol{UC}^T+\boldsymbol{U}_{X⊥} \boldsymbol{P}_{X⊥}^T+\boldsymbol{E}_y $$ 式中,组学数据集 \(\boldsymbol{X}\) 和\(\boldsymbol{Y}\)各自被分为三部分:1,两个组学相关的部分,其中,矩阵\(\boldsymbol{T}\)和\(\boldsymbol{U}\)为该部分的隐变量(成分), 而\(\boldsymbol{W}\)和\(\boldsymbol{C}\)分别为\(\boldsymbol{T}\)和\(\boldsymbol{U}\)的载荷; 2,两个组学各自特有的部分,其中,隐变量\(\boldsymbol{T}_{Y⊥}\)和\(\boldsymbol{U}_{X⊥}\)分别代表\(\boldsymbol{X}\),\(\boldsymbol{Y}\)特有的部分,与\(\boldsymbol{Y}\),\(\boldsymbol{X}\)线性无关; 3,残差项\(\boldsymbol{E}_X\),\(\boldsymbol{E}_Y\)(图3.7)。
本项目结果图见结果文件
08_O2PLS分析/本部分包含以下结果:
├── 06_O2PLS分析
├── barplot.svg [依据载荷筛选出的重要基因和代谢物]
├── loading.svg [载荷图]
├── result.html [网页版O2PLS分析结果]
├── score.svg [隐变量得分图]
└── total.svg
图3.8 O2PLS分析
注:(a)依据载荷绝对值筛选出的重要的基因和代谢物图;(b)载荷图,代表载荷对不同主成分的贡献;(c)隐变量散点图,表示样本在PC1和PC2轴上隐变量的贡献度。
3.3.4 PLS-DA分析
上述三个模型均为无监督学习(Unsupervised Learning),无法将分组信息纳入到模型中。而PLS-DA模型[20]属于监督学习(Supervised Learning)。监督学习的目标是获得一个包含特征的输入并返回对目标变量的预测[21]。该模型可以同时整合转录组和代谢组(能整合多组不同类型的组学),分组信息也会被作为虚拟变量(Dummy Variable ,即哑变量)纳入模型中。此外,环境变量也可以纳入模型中(假定分组信息为包含分类变量n维向量;环境变量为包含连续型变量n维向量)。
本项目结果图见结果文件 09_PLS_DA分析/
本部分包含以下内容:├── 09_PLS_DA分析
├── Comp.svg [载荷图]
├── Diablo.svg [两组学对应关系图]
├── auroc.svg [auc-roc曲线图]
├── biplot.svg [双序图]
├── loading.svg [载荷图]
└── plotVar.svg [相关性圈图]
(a)
(b)
(c)
(d)
图3.9 PLS-DA分析
注:(a)PLS-DA分析双序图,该图具有两套原点和单位向量方向相同的坐标系。图中散点代表隐变量而箭头代表载荷。散点表示样本在PC1和PC2轴上的隐变量的贡献度,对应Component坐标系(左下坐标轴);从原点出发的箭头的端点表示载荷值,对应Eigenvector of Component坐标系(右上)。其生物学意义的解析见结题报告3.3.5 重要的图的解读;(b)隐变量散点图,其中Block1和Block2分别代表转录组和代谢组,散点表示样本在PC1和PC2轴上隐变量的贡献度;(c)依据载荷绝对值筛选出的重要的基因和代谢物图;(d)相关性圈图,代表载荷的分布情况。3.3.5 重要的图的解读
本流程中涉及到的多元统计方法包括CCA, PLS, PLS-DA, O2PLS等。这些方法都将原始的数据分为隐变量(成分)及其所对应的载荷。双序图(biplot,有时也被译为双标图,图3.10(a))是一种集成图形,能同时展示隐变量和载荷的信息[22],注意在双序图中同时存在两套坐标系。在本流程结果的双序图中,用点表示样本,每个点在PC1和PC2轴上的投隐变量对隐变量的贡献度。从原点出发的箭头的端点表示载荷值。
这里重点介绍双序图的生物学意义。对于图中的任意两个样本点,从原点出发到样本点之间的夹角为锐角时,代表两个样本点所对应的样本具有相关基因表达谱(或代谢物丰度谱)。例如图A中SF268,SF295, SF539, SNB19, U251等在双序中相互接近,这些样本来源于同一个细胞系并具有相似的基因表达模式(图3.10(b))。夹角为钝角的具有相反的表达模式。若夹角接近直角,则表示这两个样本的表达谱不相关。两个箭头之间的夹角表示箭头所对应的基因(或代谢物)在不同样本的表达量的相关性。例如图中HEXB, WASL,GNS,PCBP4,DOCK7的夹角均为锐角,这些基因在不同样本呈现相似的表达量。在CNS和Melanoma两个细胞系的表达量高于Leikmeia。点和箭头之间的夹角代表表达量(或丰度)高低。角度越接近则该点所对应的样本中该基因(或代谢物)的表达量(或丰度)越高,例如样本SF268和基因PBCE1。反之,角度为钝角则意味着该点所对应的样本中该基因(或代谢物)的表达量(或丰度)较低,例如样本SF268和基因DPPA5[5],[23]。
当仅需要展示载荷时,可使用相关性圈图(Correlation Circles Plot,图3.10(c))。在该图中,使用标准差对载荷进行缩放,从而使每个处理所有变量之和为1。图中展示了一个半径为1的单位圆,如果一个箭头的端点接近单位圆(半径接近1),则可认为该基因(或代谢物)能够很好的由隐变量1和2代表;反之,如果一个箭头的端点距离单位圆较远(半径小于1),则可能需要使用额外的隐变量展示该箭头所对应的基因或代谢物。有时也会用圆点来代替箭头表示载荷。
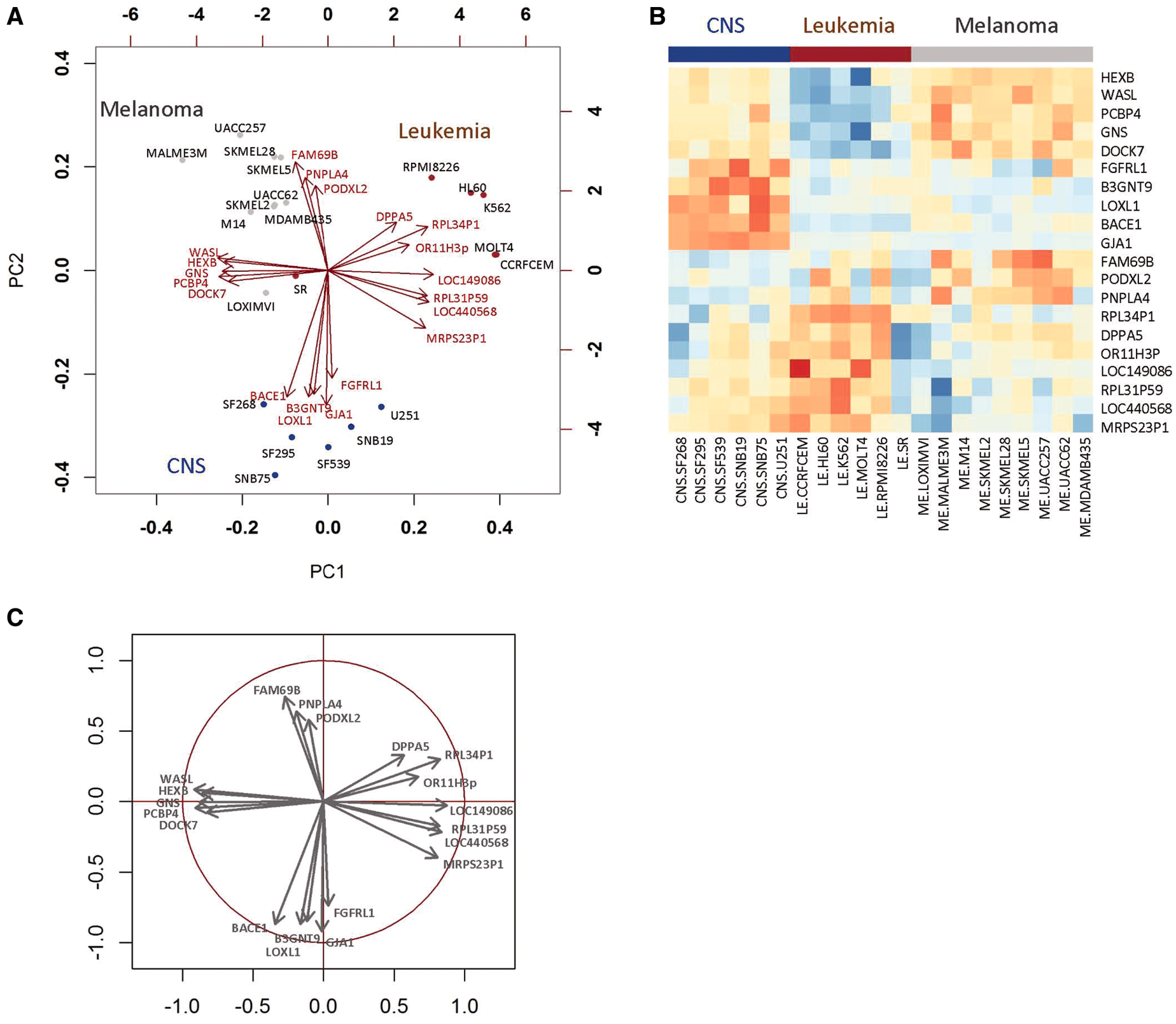
图3.10 统计模型部分的图示
注:a, 双序图;b, 相关性热图;c, 相关性圈图
(资料来源:Meng C., Zeleznik O., et
al. Dimension Reduction Techniques for the Integrative Analysis of Multi-Omics Data. Brief. Bioinform.
17, 628–641 (2016).)
四 所用软件的版本
| 软件 | 版本 |
|---|---|
| R | 4.3.1 |
| ggplot2 | 3.4.3 |
| tidyr | 1.3.0 |
| dplyr | 1.1.3 |
| pheatmap | 1.0.12 |
| ggExtra | 0.10.1 |
| pathview | 1.38.0 |
| poolr | 1.1.1 |
| mixOmics | 6.22.0 |
| OmicsPLS | 2.0.2 |
| clusterProfiler | 4.6.2 |
| patchwork | 1.1.3 |
| R2HTML | 2.3.3 |
| Python | 3.8.5 |
| Pandas | 1.1.2 |
表4.1 本流程使用软件的版本
五 参考文献
[1] Rohart F., Gautier B., et al. mixOmics: An R Package for Omics Feature Selection and Multiple Data Integration. PLOS Comput. Biol. 13, e1005752 (2017).
[2] Yan J., Risacher L., et al. Network Approaches to Systems Biology Analysis of Complex Disease: Integrative Methods for Multi-Omics Data. Brief. Bioinform. 19, 1370–1381 (2018).
[3] Kanehisa M., and Goto S. KEGG: Kyoto Encyclopedia of Genes and Genomes. Nucleic Acids Res. 28, 27–30 (2000).
[4] Butte J., Tamayo, et al. Discovering Functional Relationships Between RNA Expression and Chemotherapeutic Susceptibility using Relevance Networks. Proc. Natl. Acad. Sci. U. S. A. 97, 12182–12186 (2000).
[5] González I., Cao, et al. Visualising Associations between Paired ‘Omics’ Data Sets. BioData Min. 5, 19 (2012).
[6] Luo W., and Brouwer C. Pathview: An R/Bioconductor Package for Pathway-Based Data Integration and Visualization. Bioinformatics 29, 1830–1831 (2013).
[7] Kanehisa M., Furumichi M., et al. KEGG: New Perspectives on Genomes, Pathways, Diseases and Drugs. Nucleic Acids Res. 45, D353–D361 (2017).
[8] Koonin V. Orthologs, Paralogs, and Evolutionary Genomics. Annu. Rev. Genet. 39, 309–338 (2005).
[9] Khatri P., Sirota M. and Butte A. J. Ten Years of Pathway Analysis: Current Approaches and Outstanding Challenges. PLoS Comput. Biol. 8, e1002375 (2012).
[10] Goeman J., and Bühlmann P. Analyzing Gene Expression Data in Terms of Gene Sets: Methodological Issues. Bioinforma. Oxf. Engl. 23, 980–987 (2007).
[11] Subramanian A., Tamayo P., et al. Gene Set Enrichment Analysis: A knowledge-Based Approach For Interpreting Genome-Wide Expression Profiles. Proc. Natl. Acad. Sci. 102, 15545–15550 (2005).
[12] Wu T., Hu E., et al. ClusterProfiler 4.0: A Universal Enrichment Tool for Interpreting Omics Data. The Innovation 2, 100141 (2021).
[13] Canzler S., and Hackermüller J. multiGSEA: a GSEA-Based Pathway Enrichment Analysis for Multi-Omics Data. BMC Bioinformatics 21, 561 (2020).
[14] Cinar O., and Viechtbauer W. The poolr Package for Combining Independent and Dependent p Values. J. Stat. Softw. 101, (2022).
[15] Meng C., Zeleznik O., et al. Dimension Reduction Techniques for the Integrative Analysis of Multi-Omics Data. Brief. Bioinform. 17, 628–641 (2016).
[16] Hong S., Chen X., et al. Canonical Correlation Analysis for RNA-seq Co-Expression Networks. Nucleic Acids Res. 41, e95 (2013).
[17] Witten M., and Tibshirani J. Extensions of Sparse Canonical Correlation Analysis with Applications to Genomic Data. Stat. Appl. Genet. Mol. Biol. 8, 28 (2009).
[18] Krishnan A., William J., et al. H. Partial Least Squares (PLS) Methods for Neuroimaging: A Tutorial and Review. NeuroImage 56, 455–475 (2011).
[19] Szymanski J., Brotman Y., et al. Linking Gene Expression and Membrane Lipid Composition of Arabidopsis. Plant Cell 26, 915–928 (2014).
[20] Singh A., Shannon C., et al. DIABLO: An Integrative Approach for Identifying Key Molecular Drivers from Multi-Omics Assays. Bioinformatics 35, 3055–3062 (2019).
[21] Eraslan G., Avse Ž., et al. Deep Learning: New Computational Modelling Techniques for Genomics. Nat. Rev. Genet. 20, 389–403 (2019).
[22] Gabriel R. The Biplot Graphic Display of Matrices with Application to Principal Component Analysis. Biometrika 58, 453–467 (1971).
[23] Oyedele F., and Lubbe S. The Construction of a Partial Least-Squares Biplot. J. Appl. Stat. 42, 2449–2460 (2015).
