微科盟脂质组分析结题报告
一、 概述
脂质(Lipids)是一类疏水性或两性小分子,包括八大类(LIPID MAPS系统命名):脂肪酸类(如亚油酸、花生酸等)、甘油酯类(如TG、DG等)、甘油磷脂类(如PC、PE、PG、PA等)、鞘脂类(如Cer、SM等)、固醇脂类(如固醇脂等)、糖脂类(如MGDG、SQDG等)、孕烯醇酮脂类(如COA等)和多聚乙烯类(如抗生素等)。脂质是生物体膜结构(如细胞外膜、线粒体、外泌体、内质网等亚细胞)的主要成分,同时也是信号小分子和能量物质。因此,脂质不仅参与生长发育、神经信号转导、光合作用等多种生理过程,而且脂质代谢紊乱还与各种病理发生与发展有关,如心血管代谢综合征、肿瘤、神经退行性疾病等,以及植物的生物胁迫与非生物胁迫等应激反应密切相关。
脂质组学(Lipidomics)是一种基于高通量分析技术,系统性解析生物体脂质组成与表达变化的研究模式。脂质组学分析,可以高效地研究脂质类家族、脂质分子在各种生物过程中的改变与功能,进而阐明相关的生物活动过程与机制。
二、 实验流程
主要步骤包括:样品制备、QC制备、样品LC-MS/MS质谱分析和数据分析等。
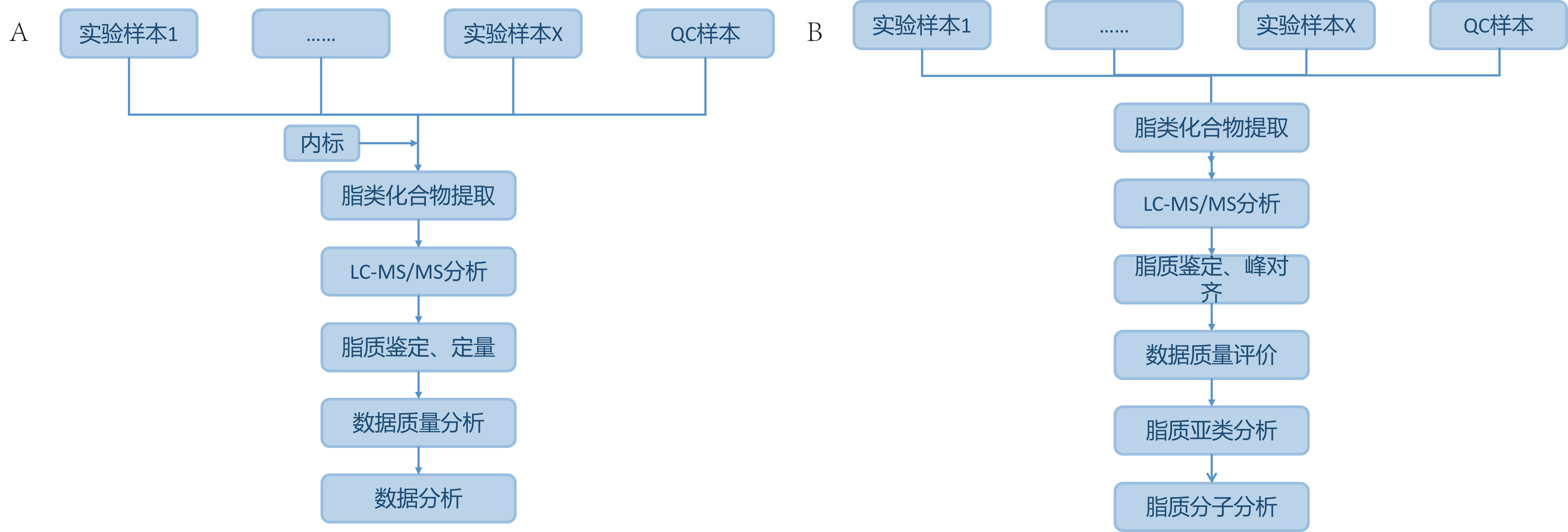
图1 技术路线(A图为绝对定量技术路线;B图为相对定量路线)
三、 实验仪器和试剂
Q-Exactivate Plus质谱仪(Thermo Scientific)
UHPLC Nexera LC-30A超高效液相色谱仪(SHIMADZU)
低温高速离心机(Eppendorf 5430R)
色谱柱:Waters,ACQUITY UPLC CSH C18,1.7 μm,2.1 mm × 100 mm column
乙腈(Thermo Fisher)
异丙醇(Thermo Fisher)
甲醇(Thermo Fisher)
14种同位素内标(Internal Standards:SPLASH LIPIDOMIX MASS SPRCSTANDARD,AVANTI,330708-1EA)
四、实验方法
4.1 样品信息
├── sample_info.txt [样本信息]
└── Lipidomics.xlsx [各脂质详细信息]
质控样本(QC)的制备:等量取各组样本混合为QC。QC样本不仅用于测定进样前仪器状态及平衡色谱-质谱系统,也穿插在待测样本检测过程中,用于评价整个实验过程中系统稳定性。
下表为样品信息一览表。
表1 分组信息一览表
| samples | category |
|---|---|
| high_1 | high |
| high_2 | high |
| high_3 | high |
| high_4 | high |
| high_5 | high |
| high_6 | high |
| high_7 | high |
| high_8 | high |
| high_9 | high |
| high_10 | high |
| middle_1 | middle |
| middle_2 | middle |
| middle_3 | middle |
| middle_4 | middle |
| middle_5 | middle |
| middle_6 | middle |
| middle_7 | middle |
| middle_8 | middle |
| middle_9 | middle |
| middle_10 | middle |
| low_1 | low |
| low_2 | low |
| low_3 | low |
| low_4 | low |
| low_5 | low |
| low_6 | low |
| low_7 | low |
| low_8 | low |
| low_9 | low |
| low_10 | low |
4.2 数据分析流程
采用LipidSearch对脂质分子及内标脂质分子进行峰识别、峰提取、脂质鉴定(二级鉴定)等处理。主要参数为:precursor tolerance: 5ppm,product tolerance: 5ppm,product ion threshold: 5%。
对LipidSearch提取得到的数据首先进行质量评价,通过后再进行数据分析。数据分析内容包括鉴定数量统计、脂质组成分析和脂质差异分析。脂质组成分析包括脂质亚类组成和脂质含量分布分析;脂质差异分析包括脂质含量、链长度、链饱和度分析;其中脂质含量变化分析又涉及整体、亚类、分子等多个维度的分析内容,详细的数据分析流程及内容如下图所示:
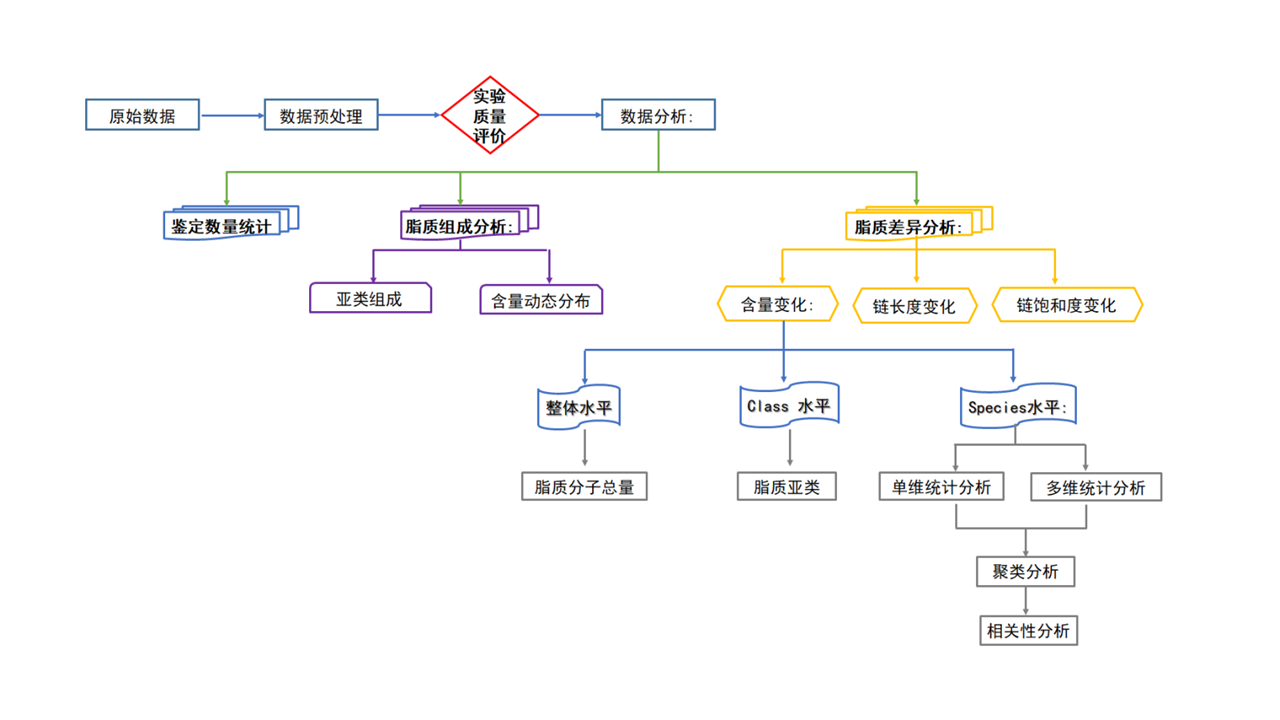
图2 脂质组分析流程
五、 数据分析
5.1 鉴定数据统计
├── Lipid_count.txt [脂质大类统计信息]
├── Lipid_count2.txt [脂质亚类统计信息]
└── sub_class_count.svg [脂质亚类统计图]
国际脂质分类和命名委员会(International Lipid Classification and Nomenclature Committee)将脂类化合物分为 8 大类型,每个类型又可以根据极性头部的不同分为不同的亚类(lipid class),每一亚类根据碳链饱和度或长度等差异分为不同的分子(lipid species),因此构成了脂类化合物大类-亚类-分子三级分类。
本实验正、负离子模式鉴定到的样本中的脂质化合物数量见表 2,具体结果见Lipidomics 表.xlsx。
表2 脂质亚类和分子鉴定数量
| Lipid class | Lipid species |
|---|---|
| 37 | 1658 |
正、负离子模式鉴定到的脂质亚类(lipid class)以及各类中鉴定到的脂质分子(lipid species)数量的统计结果,见图3。
图3 脂质亚类和脂质分子数量统计图
注:图中横坐标表示检测到的各脂质亚类,纵坐标是亚类下的脂质分子数目。
5.2 脂质组成分析
├── fig5.svg [脂质含量动态分布范围图]
├── fig7.svg [脂质分子总含量图]
├── *.svg [脂质亚类组成(*为group name)]
└── mfuzz_files [样本信息]
├── mfuzz_*_both.svg [样本信息]
└── mfuzz_*_line.svg [鉴定到的脂质分子在各组样本的变化趋势(*为2-10的数字)]
样本中脂质的类别及其比例,即脂质组成。脂质组成分析是脂质数据分析的主要内容之一。一方面,脂质的组成具有样本特异性,不同类型的样本,如细胞膜、线粒体、内质网等,稳态下所包含的脂质类别及比例是不同的1,2。
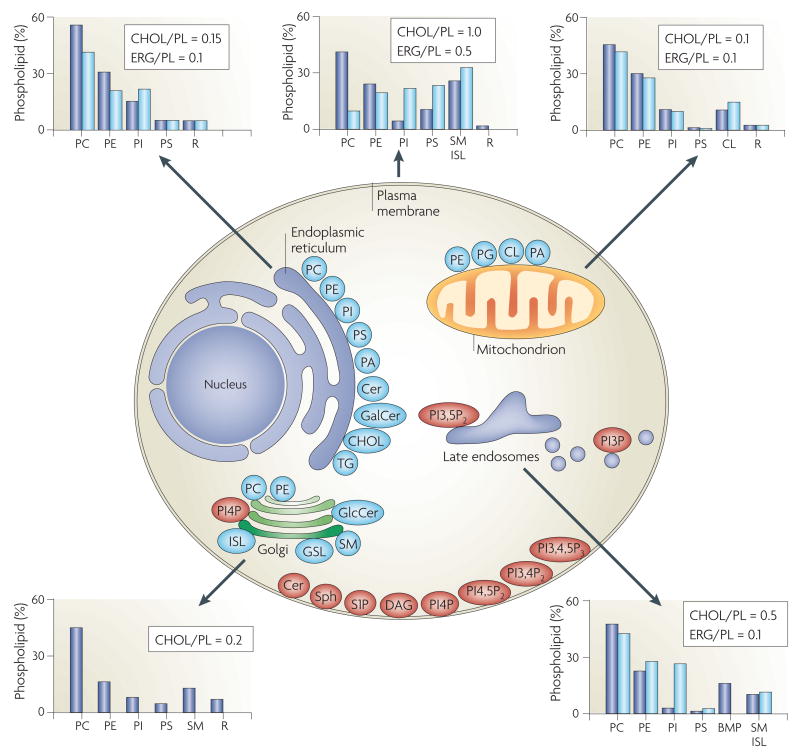
另一方面,在不同的处理条件下或生物过程中,脂质组成也会发生相应的改变,进而导致膜的生物物理特性及其功能发生变化。通过脂质组成分析,可以从整体上考察样本的主要脂质类别及其含量。
下图展示的是脂质组成分析结果: 各组样本的脂质亚类组成以环形图进行展示,如图4所示。一张环形图对应一组样本。排在前几位、比例较高的脂质亚类,为样本的主要脂质组成成分。后续分析可以将样本的主要脂质组成与文献已报道的数据进行比较,反映样本的组成和状态的变化3,4。
图4 脂质亚类组成
注:因为各类脂质分子含量差异巨大,所以对脂质含量进行log10转换后进行绘图。
图5展示的是脂质的含量动态分布范围:含量动态分布范围可以考察各组样本中含量最低、最高的脂质分子,以及脂质含量跨度范围的变化。脂质亚类的含量跨度分布,可能与不同脂质分子的贡献度及其在膜室或膜过程中的利用频率等有关5
图5 脂质含量动态分布范围
注:图中每个点代表一个脂质分子,按照含量从低到高进行排序。纵坐标是各脂质分子含量的对数值。含量最低和最高的脂质分子名称分别被标注在图中的左下角和右上角。不同的组别以不同的颜色来区分。
采用趋势聚类分析对所有鉴定到的脂质分子在各组的表达模式及变化趋势进行展示。本项目采用Mfuzz软件的fuzzy c-means(FCM)算法进行分析,根据所有脂质分子的表达趋势分为不同的表达模块,结果如图6展示。(
图6 鉴定到的脂质分子在各组样本的变化趋势
注:横坐标代表不同组别,纵坐标表示均一化之后的表达量变化。每一个Cluster的线条指表达趋势的一类代谢物。
5.3 脂质差异分析
├── 03.01Content_change_analysis
│ ├── Bubble_files
│ │ └── Bubble.*vs*.svg [差异脂质分子气泡图(*为group name)]
│ ├── cir_files
│ │ └── *vs*.cir.svg [差异脂质分子和弦图(*为group name)]
│ ├── cor_files
│ │ ├── Cor.*vs*.svg [差异脂质分子相关性聚类热图(*为group name)]
│ │ └── Cor.*vs*.top20.svg [top20差异脂质分子相关性聚类热图(*为group name)]
│ ├── fig8_files
│ │ └── fig8_*-value.svg [脂质分子亚类含量(*为脂质分子亚类名称)]
│ ├── heatmap_files
│ │ ├── group*vs*_pheatmap.png [差异脂质分子聚类热图(*为group name)]
│ │ └── group*vs*.pheatmap_top.png [top20差异脂质分子聚类热图(*为group name)]
│ ├── net_files
│ │ └── *vs*.net.svg [差异脂质分子网络图(*为group name)]
│ ├── volcano_plot_files
│ │ ├── fig9_*vs*.svg [差异脂质分子火山图(*为group name)]
│ │ ├── fig10_*vs*.svg [差异脂质分子火山图(该图标准差异脂质分子名称,*为group name)]
│ │ └── fig10_*vs*.lipid_name.txt [差异脂质分子名称列表(*为group name)]
│ ├── fig11_*vs*.svg [差异脂质分子在比较组的表达量(*为group name)]
│ ├── fig12_*vs*.svg [PCA 得分图(*为group name)]
│ ├── fig13_*vs*.svg [3D PCA 得分图(*为group name)]
│ ├── fig14_*vs*.svg [PLS-DA 得分图(*为group name)]
│ ├── fig15_*vs*.svg [OPLS-DA 得分图(*为group name)]
│ ├── OPLS-DA.R2X.*vs*.txt [OPLS-DA模型参数(*为group name)]
│ ├── pca.R2X.*vs*.txt [pca模型参数(*为group name)]
│ ├── PLS-DA.R2X.*vs*.txt [PLS-DA模型参数(*为group name)]
│ ├── venn.svg [差异脂质分子韦恩图]
│ └── vip_select.*vs*.txt [显著性差异脂质分子列表(*为group name)]
├── 03.02Chain_length_analysis
│ └── *vs*.length.svg [显著性差异脂质分子链长度统计图(*为group name)]
└── 03.03Chain_saturation_analysis
└── *vs*.unsaturation.svg [显著性差异脂质分子不饱和度统计图(*为group name)]
5.3.1 脂质含量变化分析
不同于氨基酸、核苷酸等极性代谢物,脂质的结构具有多样性,其具有大类、亚类和分子三个层次,以甘油酯(GL)类为例:所有甘油和脂肪酸(包括饱和脂肪酸和不饱和脂肪酸)经酯化所生成的酯类,均属于甘油酯大类(GL)。根据甘油酯结合的脂肪酸链的数量不同,该大类又可分为甘油结合1个脂肪酸的甘油单酯(MG),结合2个脂肪酸的甘油二酯(DG)等,即MG、DG等为甘油酯(GL)的亚类。而MG亚类,可根据其具体的脂肪酸链的长度、饱和度、结合位置等,进一步分为MG(16:0/0:0/0:0)、MG(0:0/16:0/0:0)等脂质分子,如下:
| Lipids Category | Lipids Class | Lipids Species |
|---|---|---|
| Glycerolipids(GL) | Monoradylglycerols(MG) | MG(16:0/0:0/0:0) |
| MG(0:0/16:0/0:0) | ||
| MG(20:0/0:0/0:0) | ||
| ... | ||
| Diradylglycerols(DG) | DG(16:0/16:0/0:0) | |
| DG(18:0/18:0/0:0) | ||
| DG(18:1/16:0/0:0) | ||
| ... | ||
| Triradylglycerols(TG) | TG(16:0/16:0/18:2) | |
| TG(18:0/18:0/18:0) | ||
| TG(18:1/18:1/18:1) | ||
| ... |
不同的脂质结构层次,对应不同的脂质功能层次。例如,脂质功能研究,通常更多地关注亚类层面(class)的变化,因为脂质往往以亚类集团发挥功能,目前尚难以明确区分同一亚类下单个脂质分子的功能。而标志物研究,则通常关注单个脂质分子(species)的表达水平及其诊断能力。因此,脂质含量分析涉及整体、亚类、分子三个水平,有助于更为系统、全面的揭示脂质的组间含量差异。
5.3.1.1 整体水平
将同一个样本中所有定量到的脂质分子的含量进行加合,即该样本的脂质分子总含量。然后可以对不同组别样本的总含量进行比较,结果如图7所示(
图7 脂质分子总含量
注:横坐标表示不同的组别,用不同颜色来区别。纵坐标表示不同组别的脂质分子总含量。
5.3.1.2 Class 水平
与氨基酸、核苷酸等极性代谢产物不同,脂质的功能研究主要是以亚类为单位进行的6。不同的脂质亚类,生物功能具有一定差异,例如:在脂质亚类层面,神经酰胺(Cer)与鞘磷脂(SM)虽都属于鞘脂大类,可在酶的作用下相互转化,但功能却不同: Cer 作为第二信使,对于细胞的生长、增殖、凋亡具有重要的调节作用,而 SM 与胆固醇和鞘糖脂相互作用促使脂筏的形成,参与细胞信号传导、脂质和蛋白质的分选以及细胞膜的转运7,8,9。目前已知的部分脂质亚类的部分功能如下图所示10:
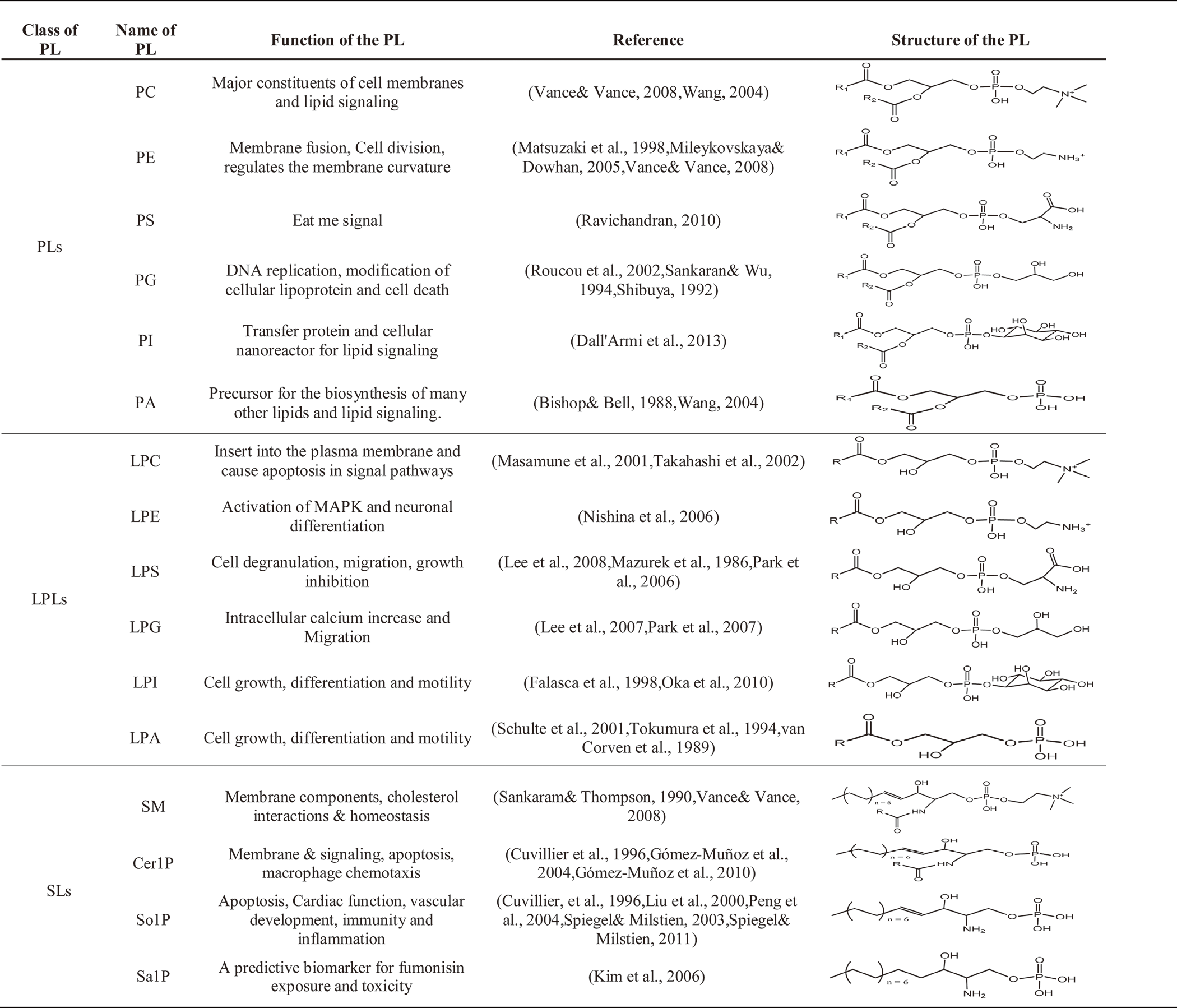
脂质亚类含量变化可反映脂质功能的变化。因此,通过比较不同样本中脂质亚类的表达变化,能够筛选出可能参与相关生物过程的重要脂质亚类,并结合该脂质亚类的功能对相关生物过程或表型进行解释。 为方便查看,对每个脂质亚类进行单独绘图,以直观了解脂质亚类在不同分组间的含量差异。
图8 脂质亚类含量
注:横坐标表示样本组别,不同组别用不同颜色来区别,纵坐标表示脂质亚类的含量。图上方横跨两组之间的数值为两组直接进行比较的p值。
5.3.1.3 Species 水平
Species 水平的分析内容与非靶代谢组的分析类似,包括多维和单维统计分析。多维统计分析是从总体水平反映组内的变异度和组间差异,同时结合单维统计分析,筛选组间显著性差异代谢物。这些显著性差异代谢物可能是潜在的生物标志物或功能分子。通过相关性分析,还可对脂质的共调控关系做进一步的挖掘。
(1)单变量统计分析
单变量统计分析方法是最常用的统计分析方法之一。在进行两组样本间的差异分析时,常用的单变量统计分析方法包括变异倍数分析(Fold Change Analysis, FC Analysis)、 T 检验/非参检验等,在该项目中使用T检验或Wilcoxon检验进行差异显著性分析。
基于单变量分析,对所有检测到的脂质分子进行了差异分析,并将分析结果以火山图的形式来进行展示,结果如图9所示。
图9 火山图
注:图中横坐标表示 log2 转换后的差异表达倍数值,纵坐标表示 log10 转换后的 Pvalue 值,点表示脂质分子。图中玫红色为显著上调脂质分子,绿色为显著下调脂质分子,灰色为统计学不显著脂质分子。
在图9的基础上,挑选表达变化(FC)上调的 TOP 10 和下调的 TOP 10 进行标记。结果如图10所示。
图10 TOP10 脂质分子标记的火山图
注:图中横坐标表示 log2 转换后的差异表达倍数值, 纵坐标表示 log10 转换后的 P value 值,点表示脂质分子。图中玫红色为显著上调脂质分子,绿色为显著下调脂质分子,灰色为统计学不显著脂质分子。图中标记的脂质分子为显著性差异代谢物中表达变化(FC)上调的 TOP 10 和下调的 TOP 10。
为了更清楚、直观的展示差异脂质分子的表达量变化,差异的脂质分子按照所属亚类进行作图展示。以 PC 为例,差异脂质分子在示例对比组的表达量差异如图11所示。
图11 差异脂质分子在比较组的表达量
注:横坐标表示差异脂质分子,纵坐标为脂质分子的绝对含量或相对含量。
(2) 多维统计分析
a) 主成分分析(PCA)
主成分分析(Principal Component Analysis, PCA)是一种非监督的数据分析方法,它将原本鉴定到的所有脂质分子重新线性组合,形成一组新的综合变量,同时根据所分析的问题从中选取几个综合变量,使它们尽可能多地反映原有变量的信息,从而达到降维的目的。同时,对脂质进行主成分分析,还能从总体上反映样本组间和组内的变异度。因此在数据分析中,一般先采用 PCA 方法,观察组间样本的总体分布趋势和组间样本的差异度。
分别对各个比较组做 PCA 分析,以示例对比组为例进行统计分析, PCA 得分图见图12。
图12 PCA 得分图
注:图中PC1代表主成分1,PC2代表主成分2,椭圆代表95%置信区间。同一颜色的点表示组内的各个生物学重复,点的分布状态反映出的是组间、组内的差异度。
3D PCA 得分图,结果如图13所示。
图13 3D PCA 得分图
注:图中PC1代表主成分1,PC2代表主成分2,PC3代表主成分3。同一颜色的点表示组内的各个生物学重复,点的分布状态反映出的是组间、组内的差异度。
经 7-fold cross-validation(7 次循环交互验证)得到的 PCA 模型参数见表 3。R2X 越接近 1 表明模型越稳定可靠。
表3 PCA模型的评价参数
| 样品分组 | A | R2X(cum) |
|---|---|---|
| highvslow | 3 | 0.773763 |
| highvsmiddle | 3 | 0.813046 |
| lowvsmiddle | 3 | 0.770195 |
注:表中 A:表示主成分数; R2X:表示模型解释率。
b) 偏最小二乘判别分析(PLS-DA)
偏最小二乘判别分析(Partial Least Squares Discrimination Analysis, PLS-DA)是一种有监督的判别分析统计方法。该方法运用偏最小二乘回归建立脂质表达量与样品类别之间的关系模型,来实现对样品类别的预测。通过建立的判别模型,可以从数据集中筛选出与分组相关的差异脂类物质。
示例对比组的 PLS-DA 模型得分图见图14。由此可见, PLS-DA 模型能区分两组样本。
图14 PLS-DA 得分图
注: 图中P1代表主成分1,P2代表主成分2,椭圆代表95%置信区间。同一颜色的点表示组内的各个生物学重复,点的分布状态反映出的是组间、组内的差异度。
经7-fold cross-validation(7次循环交互验证)得到的模型评价参数(R2Y,Q2)列于表4。一般Q2大于0.5,表明模型稳定可靠, 0.3
表4 PLS-DA 模型的评价参数
| 样品分组 | A | R2X(cum) | R2Y(cum) | Q2(cum) |
|---|---|---|---|---|
| highvslow | 2 | 0.808 | 0.993555555555556 | 0.845333333333333 |
| highvsmiddle | 2 | 0.2543 | 0.3679 | 0.14092 |
| lowvsmiddle | 2 | 0.8072 | 0.9917 | 0.8033 |
注:表中 A:表示主成分数; R2X:表示模型对 X 变量解释率; R2Y:表示模型对Y变量的解释率; Q2:表示模型预测能力。
c) 正交偏最小二乘判别分析(OPLS-DA)
正交偏最小二乘判别分析(OPLS-DA)是一种对PLS-DA进行修正的分析方法,可以滤除与分类信息无关的噪音,提高了模型的解析能力和有效性;在OPLSDA得分图上,有两种主成分,即预测主成分和正交主成分。 OPLS-DA将组间差异最大化的反映在 t[1]上,所以从 t[1]上能直接区分组间变异,而在正交主成分 to[1]上则反映了组内的变异。
示例对比组的OPLS-DA模型得分图见图15,可见OPLS-DA模型能区分两组样本。
图15 OPLS-DA 得分图
注:图中P1代表主成分1,P2代表主成分2,椭圆代表95%置信区间。同一颜色的点表示组内的各个生物学重复,点的分布状态反映出的是组间、组内的差异度。
经 7-fold cross-validation(7 次循环交互验证)得到的模型评价参数(R2Y, Q2)列于表 5。一般 Q2 大于 0.5,表明模型稳定可靠, 0.3<Q2≤0.5,表明模型稳定性较好, Q2<0.3,表明模型可靠性较低。
表 5 OPLS-DA 模型的评价参数
| 样品分组 | A | R2X(cum) | R2Y(cum) | Q2(cum) |
|---|---|---|---|---|
| highvslow | 2 | 0.7561 | 0.9669 | 0.4196 |
| highvsmiddle | 2 | 0.799444444444444 | 0.866888888888889 | 0.445666666666667 |
| lowvsmiddle | 2 | 0.7248 | 0.8266 | 0.4166 |
注:表中A:表示主成分数;R2X:表示模型对X变量解释率;R2Y:表示模型对Y变量的解释率;Q2:表示模型预测能力。
(3)显著性差异脂质分子
OPLS-DA 模型得到的变量权重值(Variable Importance for the Projection, VIP)能够用于衡量各脂质分子的表达模式对各组样本分类判别的影响强度和解释能力,挖掘具有生物学意义的差异脂质分子。通常 VIP>1 的脂质分子被认为在模型解释中具有显著贡献。本实验以 OPLS-DA VIP>1 和 P value < 0.05 为显著性差异脂质分子筛选标准。部分显著性差异脂质分子见表6,详细信息见文件夹customer_files/03variance_analysis/03.01Content_change_analysis/vip_select.*vs*.txt。
表6 部分显著性差异脂质分子
| lips | VIP | highvslow_p | highvslow_fc | highvslow_change |
|---|---|---|---|---|
| SPH(t17:1)+H | 2.89564595713909 | 4.33003528978761e-05 | -1.34426903406456 | down |
| Cer(d34:2)+H | 2.76636372141043 | 3.01749061997166e-05 | 1.13962643360058 | up |
| Cer(d34:1)+H | 2.69214993380432 | 5.29507651635806e-05 | 1.0341753529059 | up |
| WE(14:0_17:3)+NH4 | 2.51566577687872 | 0.000741348364975215 | 1.52715657185382 | up |
| PC(34:1)+H | 2.49803274646183 | 0.00893069778518695 | 1.73591118919587 | up |
| DG(21:2e)+Na | 2.48897143740126 | 0.00144409296341941 | 2.96152003389124 | down |
| TG(6:0_6:0_23:1)+NH4 | 2.3175952060838 | 0.00151118569482734 | 0.465036286872086 | not_sign |
各对比组筛选到的显著性差异脂质分子的重叠情况,以 Venn 图的形式进行展示,结果如图 16 所示。根据 Venn 图,能够直观的看出有多少显著性差异脂质分子在不同对比组均发生变化,有多少显著性差异脂质分子仅在某一对比组发生变化,有助于筛选与生物过程相关的核心脂质分子模块。
图16 差异脂质分子韦恩图
(4)气泡图
以示例对比组为例,对本实验筛选到的显著性差异脂质分子(VIP>1, P value<0.05),以气泡图形式进行可视化展示11,如图17所示。
图17 气泡图
注:图中气泡表示显著性差异脂质分子;气泡颜色对应其-log10(pvalue)值,气泡大小对应其foldchange值。
(5) 聚类分析
为了评价差异脂质的合理性,同时更全面直观地显示样本之间的关系以及脂质在不同样本中的表达模式差异,我们利用显著性差异脂质(VIP>1, P value<0.05)的表达量对各组样本进行层次聚类(Hierarchical Clustering)。一般来说,当筛选的候选脂质合理且准确时,同组样本能够通过聚类出现在同一簇(Cluster)中。同时,聚在同一簇内的脂质具有相似的表达模式,可能在代谢过程中处于较为接近的反应步骤中。示例对比组的聚类分析结果如图18所示。
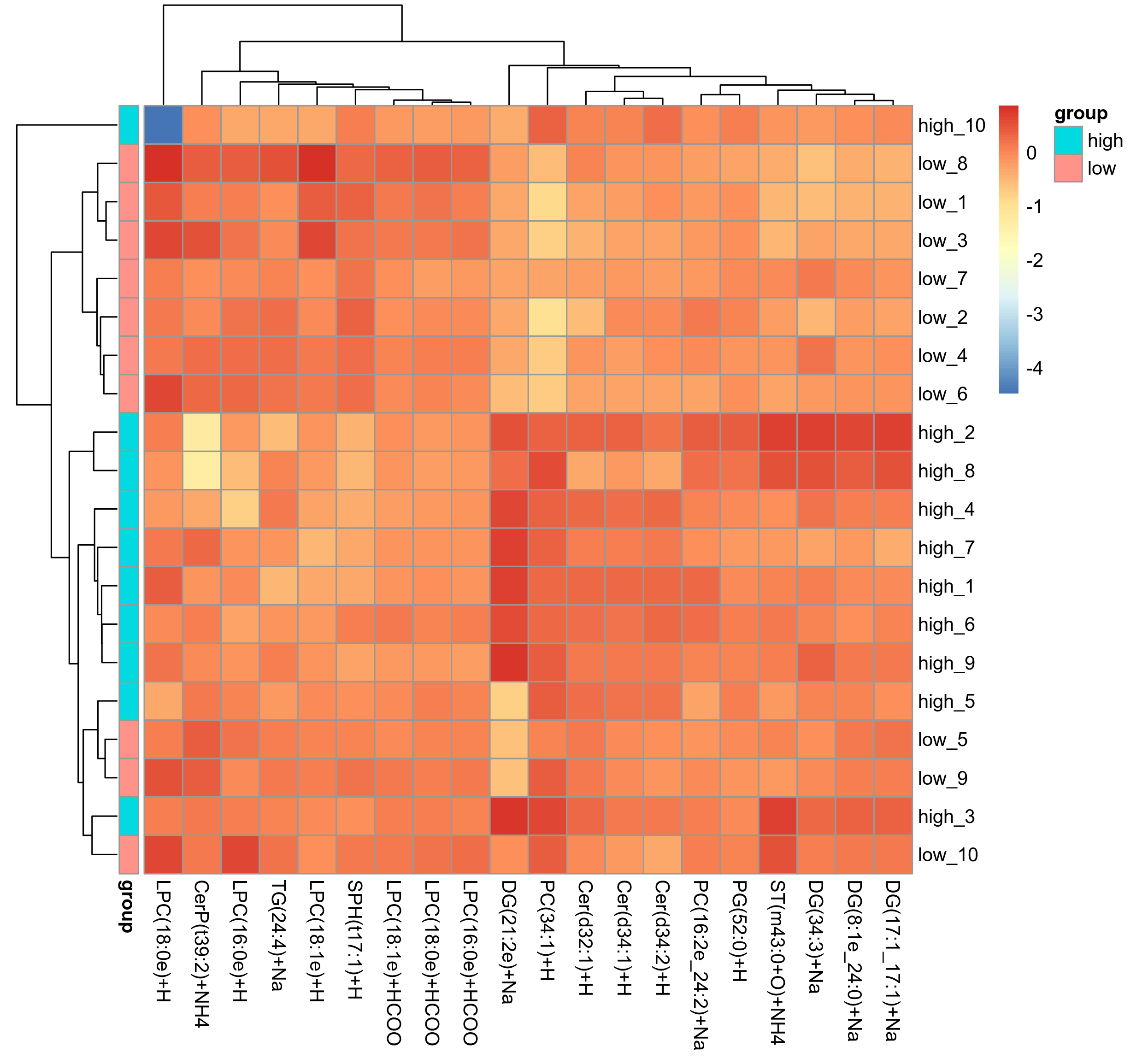
图18 聚类热图
注: 图中每列代表一个差异脂质分子(即横坐标为显著性差异表达的脂质分子),每行代表一组样品(即纵坐标为样品信息)。 不同位置的色块代表对应位置脂质分子的相对表达量,红色代表表达量相对较高,蓝色代表表达量相对较低,表达模式接近的脂质分子聚在左侧同一 cluster 下。
(6) 相关性分析
相关性分析可以帮助衡量显著性差异脂质(VIP>1, P value < 0.05)之间的代谢密切程度(metabolic proximities), 有利于进一步了解生物状态变化过程中脂质之间的相互调节关系。具有表达相关性的脂质,可能共同参与某一生物过程,即功能相关性;此外,正相关的脂质也可能表明其来源于同一合成途径,负相关表明可能被分解用于其他脂质的合成,即合成转化关系。
基于相关性分析方法,对显著性差异脂质之间的相关性进行分析。相关性分析的结果以相关性聚类热图的形式来进行可视化展示,如图19所示。
图19 相关性聚类热图
注: 红色表示正相关,蓝色表示负相关,颜色深浅与相关性系数的绝对值大小有关,即正相关或负相关的程度越高,颜色越深。
具有强正相关或负相关关系的脂质会聚在上图中的同一个 Cluster 内。后续可进一步通过对 Cluser 内的脂质亚类组成以及 Cluster 之间的相关性进行分析,分析脂质亚类之间的共调控关系,其是否具有合成转化关系或者功能相关性。
例如,脂质之间通过脂代谢酶进行相互转化,部分脂质的代谢网络图如下图所示12。因此后续可以参考文献报道或先前的研究结果对相关脂代谢酶的表达量进行检测, 揭示该生物条件下的脂质合成转化关系; 进一步,通过对脂代谢酶的功能研究, 回到基因或蛋白层面深入揭示相关的分子机理, 提高研究档次11。 若是临床样本, 可与临床参数相结合, 分析 Cluster是否具有分型或预后价值13。
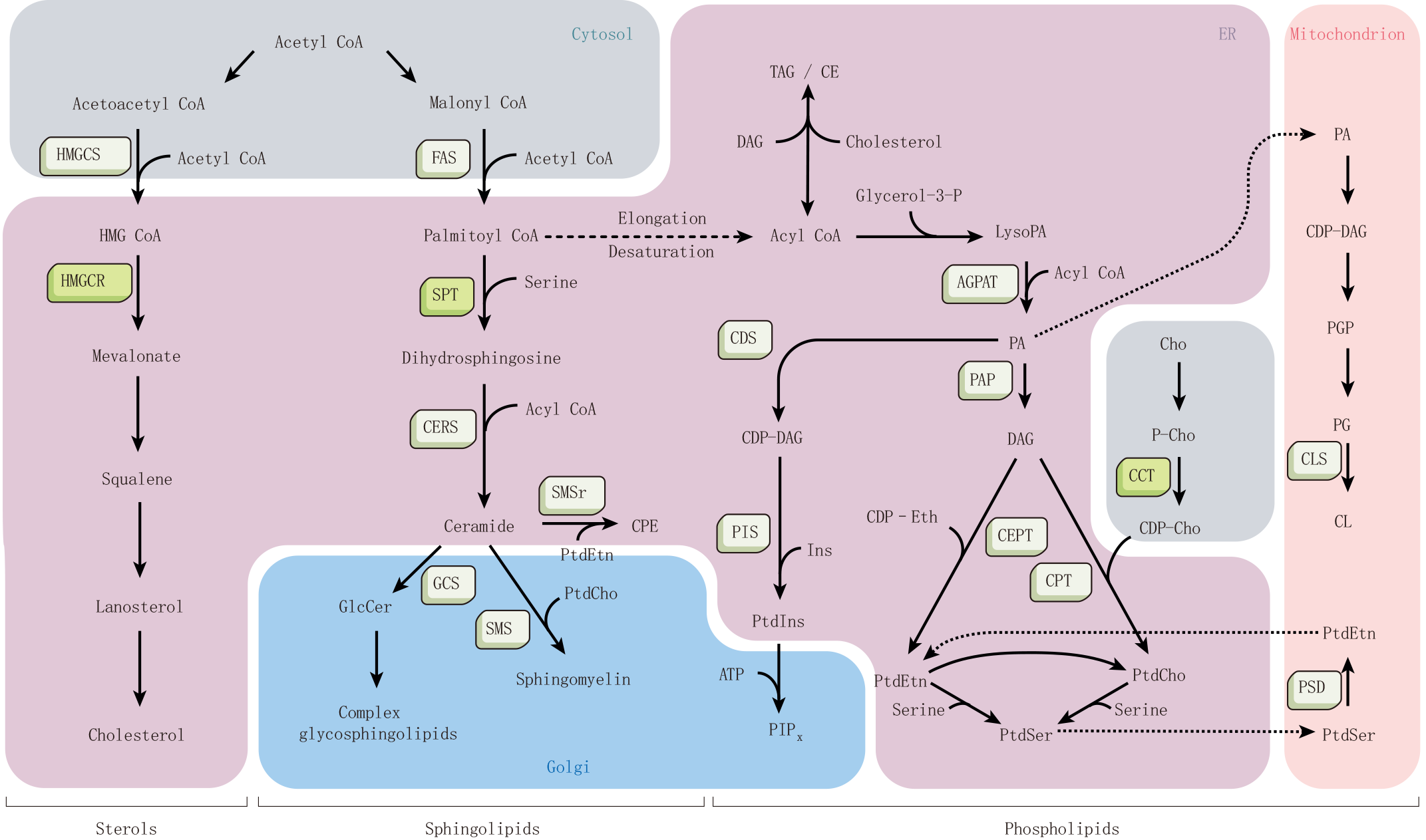
为了更直观的揭示脂质的共调节关系,脂质相关性矩阵(Lipid-lipid correlation matrix)(图19)被转换成和弦图和网络图,如图20和图21所示。和弦图和网络图均展示的是相关性系数|r|>0.8 且 p<0.05 的脂质分子对[3],此标准可根据实际的情况调整。和弦图能更好的展示脂质亚类之间的相关性,网络图能更好的展示脂质分子之间的相关性,各有优点。
图20 和弦图
注:图中内圈 link 的起点代表显著性差异脂质分子,外圈上的弧线表示脂质亚类。彩色线条表示亚类内部脂质分子的相关性,线条与亚类同色。深灰色线条表示亚类与亚类之间的相关性。
图21 网络图
若需要对网络图进行美化,可参考结果文件夹中CytoscapeQuickStart.pdf进行修改。
注:图中点代表显著性差异脂质分子,点的大小与连接度 degree 相关, degree 越大,点越大。线条的颜色代表相关性,红色表示正相关,蓝色表示负相关。线条的粗细代表相关性系数绝对值的大小,线条越粗,相关性越大。
5.3.2 链长度分析
以脂质分子 DG(16:0/16:0/0:0)为例, 链长度是该脂质分子所具有的脂肪酸链的C 原子总和,即长度为 32 个碳原子。除了脂质的含量与脂质功能有关以外,脂质的链长度也是不可忽视的一个影响因素:链长度影响细胞膜厚度,进而影响细胞膜的流动性、相关脂质转运蛋白及目标靶蛋白的活性与功能等。目前的研究表明脂质具有链特异性(chain-specific) 的生理和病理性质。 以神经酰胺(Cer) 为例14:
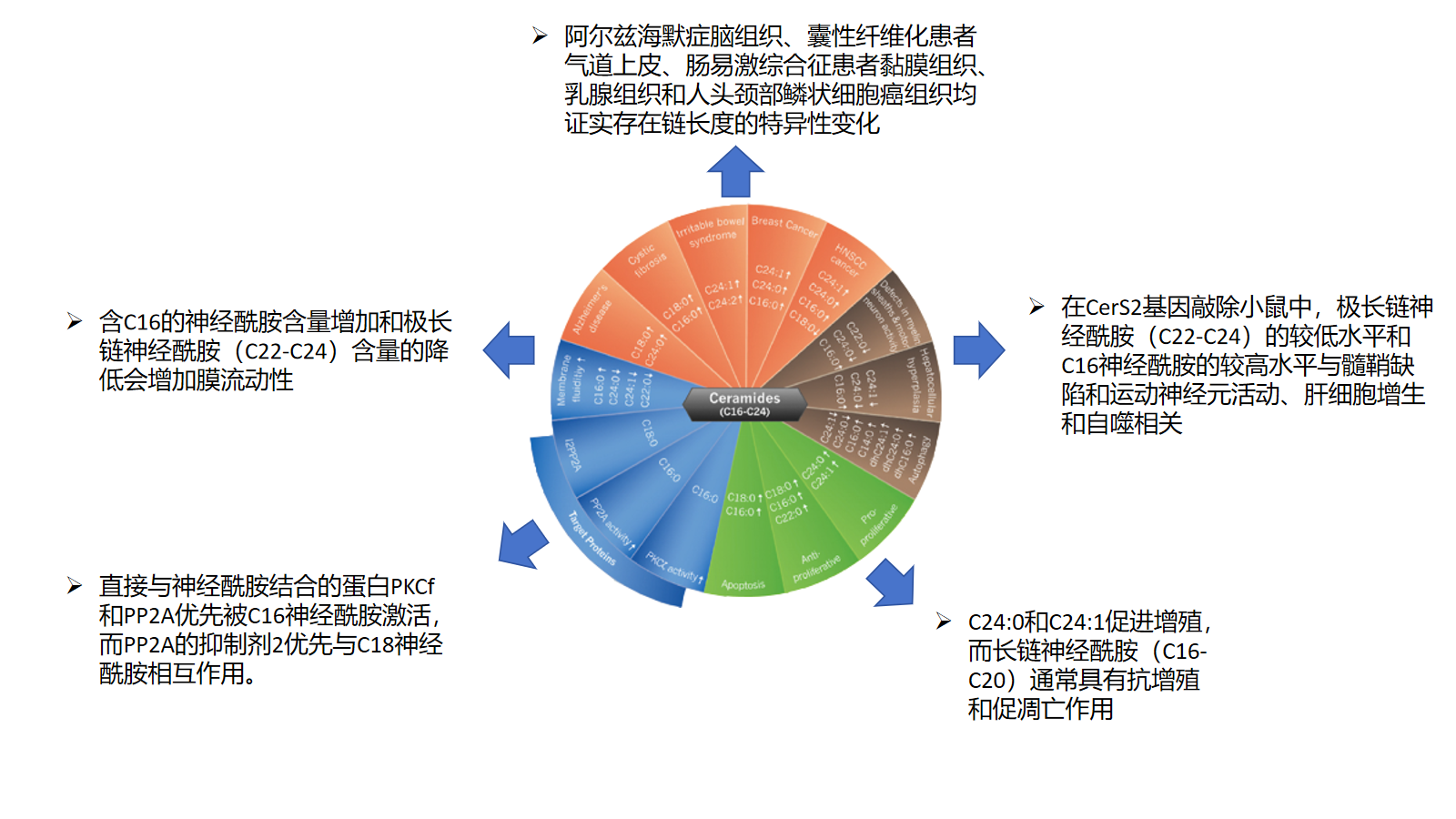
将带有相同长度的脂质分子的含量相加,统计各亚类下不同碳链长度的脂质分子含量差异。以 PC 为例,结果如图22所示。其余脂质亚类的碳链长度分布见详见customer_files/03variance_analysis/03.02Chain_length_analysis/。
建议后续结合临床参数、脂代谢酶、脂质转运蛋白、目标靶蛋白[1]等内容进行进一步研究。例如,磷酯类如 PC、 PE 等亚类是细胞膜的主要成分,其链长度的变化,会直接引起膜的流动性变化,进而影响膜的通透性、物质运输、膜蛋白的定位功能等;由于链长度的变化受到相关脂代谢酶的调控,可根据链长的变化进一步锁定可能发挥重要作用的脂代谢酶,进而深入解释相关的分子机理。
图22 碳链长度分布
注:横坐标表示不同碳链长度的脂质分子,纵坐标表示脂质分子的含量。
5.3.3 链饱和度分析
以脂质分子 TG(18:1/18:1/18:1)为例,链饱和度是该脂质分子所具有的脂肪酸链的双键数量的总和,即不饱和度为 3。除了上述的脂质含量与链长度以外,脂质的饱和度也是影响脂质功能的重要因素:双键引入扭结(kinks),降低酰基链的堆积密度,并抑制细胞膜从流体向固相凝胶状态的改变12。因此脂质饱和度通过影响细胞膜的流动性,进而影响细胞的分裂、迁移和信号转导,在疾病的发生和应激响应中起着重要的作用,同样值得引起关注和重视。目前的研究表明脂质的饱和度变化具有重要的生物意义,例如:
- 细胞膜脂质的饱和度影响膜的流动性,含有棕榈酰(C16:0)的甘油磷脂的减少,引起细胞膜流动性增加,促进了肝癌细胞的增殖和侵袭性15。
- 卵巢癌16、肺癌17、乳腺癌和肝癌等多种肿瘤干细胞中, SCD1(stearoyl-CoA desaturase-1)高表达引起相关致癌信号通路的激活,与疾病进展和不良预后密切相关。
- 内质网中饱和脂质的积累,激活未折叠蛋白反应(UPR),与 II 型糖尿病、肝衰竭以及心血管疾病的发病机制密切相关。
- 甘油三酯通过平衡脂肪酸饱和度促进低氧应激时的脂质稳态,缓解饱和脂肪酸的毒性损伤。
- 变温生物(poikilothermic organisms), 如细菌、真菌和植物,通过 homeoviscous adaptation 过程调节膜的饱和度来适应环境温度变化。
将带有相同数量不饱和键的脂质分子的含量相加,统计各亚类下不同不饱和键数目的脂质分子含量差异。以 PC 为例,结果如图23所示。其余脂质亚类的不饱和键分布详见customer_files/03variance_analysis/03.03Chain_saturation_analysis/。
建议后续结合脂代谢酶、脂质饱和度感受器(如 Mga2 等)以及信号通路等内容进行更深的机制研究。例如,磷酯类如 PC、 PE 等亚类是细胞膜的主要成分,其链饱和的变化,会直接引起膜的通透性、物质运输、膜蛋白的定位功能等;由于链饱和度的变化由相关脂代谢酶所决定,并由脂质饱和度感受器(如 Mga2 等)进行调控,可根据链饱和度的变化进一步锁定可能其重要作用的脂代谢酶或感受器,进而深入解释相关的分子机理。
图23 链饱和度分析
注:横坐标表示不饱和键数目,纵坐标表示具有相同不饱和键数目的脂质分子的含量总和。
六. 参考文献
- Membranes: a meeting point for lipids, proteins and therapies.[PMID: 18266954]
- Membrane lipids: where they are and how they behave.[PMID: 18216768]
- Lipidomics Analyses Reveal Temporal and Spatial Lipid Organization and Uncover Daily Oscillations in Intracellular Organelles.[PMID: 27161994]
- Global analysis of the yeast lipidome by quantitative shotgun mass spectrometry.[PMID: 19174513]
- Identification of key lipids critical for platelet activation by comprehensive analysis of the platelet lipidome.[PMID: 29784642]
- Lipidomics for studying metabolism.[PMID: 27469345]
- Sphingomyelin and its role in cellular signaling.[PMID: 23775687]
- Ceramide and apoptosis: exploring the enigmatic connections between sphingolipid metabolism and programmed cell death.[PMID: 21707511]
- Lipid signalling in disease.[PMID: 18216772]
- Phospholipids as cancer biomarkers: Mass spectrometry-based analysis.[PMID: 27276657]
- RNA-Seq and Mass-Spectrometry-Based Lipidomics Reveal Extensive Changes of Glycerolipid Pathways in Brown Adipose Tissue in Response to Cold.[PMID: 26628366]
- Lipid landscapes and pipelines in membrane homeostasis.[PMID: 24899304]
- An Integrated Metabolic Atlas of Clear Cell Renal Cell Carcinoma.[PMID: 26766592]
- Chain length-specific properties of ceramides.[PMID: 22133871]
- Functional lipidomics: Palmitic acid impairs hepatocellular carcinoma development by modulating membrane fluidity and glucose metabolism.[PMID: 28073184]
- Lipid Desaturation Is a Metabolic Marker and Therapeutic Target of Ovarian Cancer Stem Cells.[PMID: 28041894]
- Blockade of Stearoyl-CoA-desaturase 1 activity reverts resistance to cisplatin in lung cancer stem cells.[PMID: 28797843]
- Inhibition of fatty acid desaturation is detrimental to cancer cell survival in metabolically compromised environments.[PMID: 27042297]